책/머신러닝 완벽가이드
차원축소
광이11
2023. 12. 30. 19:28
차원 축소 개념
- 차원이(feature) 증가하면 데이터 포인트 간 거리가 기하급수적으로 멀어짐 (=희소한 구조)
- 만약 수백 개 이상 피처로 구성된 데이터 세트가 있다면 적은 차원에서 학습된 모델보다 예측 신뢰도 떨어짐
- 또한 개별 피처간 상관관계가 높을 가능성이 큼
- 선형 모델에서 입력 변수 간 상관관계가 높은 경우 다중 공산성 문제가 발생하며 결국, 모델 예측 성능이 저하됨
이러한 문제들을 해결하기 위해, 많은 다차원 피처를 줄이는 것이 차원 축소임
차원 축소 종류 2가지
피처 선택(feature selection) & 피처 추출(feature extraction)
피처 선택
- 특정 피처에 종속성이 강한 불필요한 피처 제거
- 즉, 특징을 잘 나타내는 주요 피처만 선택
피처 추출
- 기존 피처를 저차원의 중요 피처로 압축해서 추출
- 새롭게 추출된 중요 특성은 기존의 피처가 압축된 것 즉, 기존 피쳐와는 완전 다른 값
- 단순 압축 x → 또 다른 공간으로 매핑해 추출 o
- 즉, 함축적인 요약 특성 추출
- 기존 피처가 인지하기 어려웠던 잠재적 요소 추출
- ex ) 성적, 상장, 대외활동, 봉사활동, 공모전, 학업 성취도 , 모의고사 성적, 영어성적같은 피쳐를 더욱 함축
정리
- 차원축소는 단순히 데이터의 압축 아님
- 중요한 것은 차원 축소를 통해 더 데이터를 잘 설명할 수 있는 잠재적인 요소를 추출하는 것
- PCA, SVD, NMF가 대표적 (이미지, 텍스트에서 잘 활용됨)
- 이미지 데이터 : 이미지 변환, 압축 진행 → 원본 이미지보다 훨씬 적은 차원 → 과적합 영향력이 작아짐 ⇒ 예측 성능 향상
- 텍스트 문서의 숨겨진 의미 추출 → 시맨틱(semantic)한 의미, topic을 잠재 요소로 간주하고 찾음 (SVD, NMF가 semantic topic 모델링을 위한 기반 알고리즘으로 사용)
PCA
- 대표적인 차원 축소 기법
- 변수 간의 상관관계를 이용해 대표 주성분을 추출해 차원 축소
- 차원 축소 시 기존 데이터의 정보 유실을 최소화 할 것
- 가장 높은 분산을 가지는 데이터의 축을 찾아 이 축으로 차원 축소, 이것이 PCA의 주성분
- 즉, PCA는 분산이 데이터의 특성을 가장 잘 나타내는 것으로 간주
차원 축소 절차
- 가장 큰 데이터 변동성을 기반으로 첫 번째 벡터 축 생성
- 두 번째 축은 이 벡터 축에 직각이 되는 벡터를 축으로 함
- 두 번째 축과 직각이 되는 벡터를 설정하는 방식으로 축 생성
이렇게 생성된 벡터 축에 원본 데이터를 투영하면 벡터 축 개수 만큼의 차원으로 원본 데이터가 차원 축소됨
즉, PCA는 원본 데이터 피처 개수에 비해 매우 작은 주성분으로 원본 데이터의 총 변동성을 대부분 설명할 수 있는 분석법
선형대수 관점에서 본 PCA
★ 입력 데이터의 공분산 행렬이 고유벡터와 고유값으로 분해될 수 있으며, 이렇게 분해된 고유벡터를 이용해 입력 데이터를 선형 변환하는 방식이 PCA라는 것임
PCA STEP
- 입력 데이터 세트의 공분산 행렬을 생성
- 공분산 행렬의 고유벡터와 고유값을 계산
- 고유값이 가장 큰 순으로 K개(PCA 변환 차수만큼)만큼 고유벡터를 추출
- 고유값이 가장 큰 순으로 추출된 고유벡터를 이용해 새롭게 입력 데이터를 변환
붓꽃 데이터로 PCA 변환을 위한 데이터 로딩 및 시각화
from sklearn.datasets import load_iris
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
# 사이킷런 내장 데이터 셋 API 호출
iris = load_iris()
# 넘파이 데이터 셋을 Pandas DataFrame으로 변환
columns = ['sepal_length','sepal_width','petal_length','petal_width']
irisDF = pd.DataFrame(iris.data , columns=columns)
irisDF['target']=iris.target
irisDF.head(3)
#setosa는 세모, versicolor는 네모, virginica는 동그라미로 표현
markers=['^', 's', 'o']
#setosa의 target 값은 0, versicolor는 1, virginica는 2. 각 target 별로 다른 shape으로 scatter plot
for i, marker in enumerate(markers):
x_axis_data = irisDF[irisDF['target']==i]['sepal_length']
y_axis_data = irisDF[irisDF['target']==i]['sepal_width']
plt.scatter(x_axis_data, y_axis_data, marker=marker,label=iris.target_names[i])
plt.legend()
plt.xlabel('sepal length')
plt.ylabel('sepal width')
plt.show()

#평균이 0, 분산이 1인 정규 분포로 원본 데이터를 변환
from sklearn.preprocessing import StandardScaler
# Target 값을 제외한 모든 속성 값을 StandardScaler를 이용하여 표준 정규 분포를 가지는 값들로 변환
iris_scaled = StandardScaler().fit_transform(irisDF.iloc[:, :-1])
iris_scaled.shape
[output]
(150, 4)
#PCA 변환 수행
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
#fit( )과 transform( ) 을 호출하여 PCA 변환 데이터 반환
pca.fit(iris_scaled)
iris_pca = pca.transform(iris_scaled)
print(iris_pca.shape)
[output]
(150, 2)
# PCA 환된 데이터의 컬럼명을 각각 pca_component_1, pca_component_2로 명명
pca_columns=['pca_component_1','pca_component_2']
irisDF_pca = pd.DataFrame(iris_pca, columns=pca_columns)
irisDF_pca['target']=iris.target
irisDF_pca.head(3)
#PCA로 차원 축소된 피처들로 데이터 산포도 시각화
#setosa를 세모, versicolor를 네모, virginica를 동그라미로 표시
markers=['^', 's', 'o']
#pca_component_1 을 x축, pc_component_2를 y축으로 scatter plot 수행.
for i, marker in enumerate(markers):
x_axis_data = irisDF_pca[irisDF_pca['target']==i]['pca_component_1']
y_axis_data = irisDF_pca[irisDF_pca['target']==i]['pca_component_2']
plt.scatter(x_axis_data, y_axis_data, marker=marker,label=iris.target_names[i])
plt.legend()
plt.xlabel('pca_component_1')
plt.ylabel('pca_component_2')
plt.show()

#각 PCA Component별 변동성 비율*
print(pca.explained_variance_ratio_)
[output]
[0.72962445 0.22850762]
#원본 데이터와 PCA 변환된 데이터 기반에서 예측 성능 비교
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score
import numpy as np
rcf = RandomForestClassifier(random_state=156)
scores = cross_val_score(rcf, iris.data, iris.target,scoring='accuracy',cv=3)
print('원본 데이터 교차 검증 개별 정확도:',scores)
print('원본 데이터 평균 정확도:', np.mean(scores))
[output]
원본 데이터 교차 검증 개별 정확도: [0.98 0.94 0.96]
원본 데이터 평균 정확도: 0.96
pca_X = irisDF_pca[['pca_component_1', 'pca_component_2']]
scores_pca = cross_val_score(rcf, pca_X, iris.target, scoring='accuracy', cv=3 )
print('PCA 변환 데이터 교차 검증 개별 정확도:',scores_pca)
print('PCA 변환 데이터 평균 정확도:', np.mean(scores_pca))
[output]
PCA 변환 데이터 교차 검증 개별 정확도: [0.88 0.88 0.88]
PCA 변환 데이터 평균 정확도: 0.88
신용카드 데이터 세트 PCA 변환
# header로 의미없는 첫행 제거, iloc로 기존 id 제거
import pandas as pd
pd.set_option('display.max_columns', 30)
df = pd.read_excel('pca_credit_card.xls', header=1, sheet_name='Data').iloc[:,1:]
print(df.shape)
df.head(3)
df.rename(columns={'PAY_0':'PAY_1','default payment next month':'default'}, inplace=True)
y_target = df['default']
X_features = df.drop('default', axis=1)
#피처간 상관도 시각화
import seaborn as sns
import matplotlib.pyplot as plt
%matplotlib inline
corr = X_features.corr()
plt.figure(figsize=(14,14))
sns.heatmap(corr, annot=True, fmt='.1g')
plt.show()

from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
#BILL_AMT1 ~ BILL_AMT6까지 6개의 속성명 생성
cols_bill = ['BILL_AMT'+str(i) for i in range(1, 7)]
print('대상 속성명:', cols_bill)
# 2개의 PCA 속성을 가진 PCA 객체 생성하고, explained_variance_ratio_ 계산을 위해 fit( ) 호출
scaler = StandardScaler()
df_cols_scaled = scaler.fit_transform(X_features[cols_bill])
pca = PCA(n_components=2)
pca.fit(df_cols_scaled)
print('PCA Component별 변동성:', pca.explained_variance_ratio_)
[output]
대상 속성명: ['BILL_AMT1', 'BILL_AMT2', 'BILL_AMT3', 'BILL_AMT4', 'BILL_AMT5', 'BILL_AMT6']
PCA Component별 변동성: [0.90555253 0.0509867 ]
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import cross_val_score
rcf = RandomForestClassifier(n_estimators=300, random_state=156)
scores = cross_val_score(rcf, X_features, y_target, scoring='accuracy', cv=3 )
print('CV=3 인 경우의 개별 Fold세트별 정확도:',scores)
print('평균 정확도:{0:.4f}'.format(np.mean(scores)))
[output]
CV=3 인 경우의 개별 Fold세트별 정확도: [0.8083 0.8196 0.8232]
평균 정확도:0.8170
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
# 원본 데이터셋에 먼저 StandardScaler적용
scaler = StandardScaler()
df_scaled = scaler.fit_transform(X_features)
# 6개의 Component를 가진 PCA 변환을 수행하고 cross_val_score( )로 분류 예측 수행.
pca = PCA(n_components=6)
df_pca = pca.fit_transform(df_scaled)
scores_pca = cross_val_score(rcf, df_pca, y_target, scoring='accuracy', cv=3)
print('CV=3 인 경우의 PCA 변환된 개별 Fold세트별 정확도:',scores_pca)
print('PCA 변환 데이터 셋 평균 정확도:{0:.4f}'.format(np.mean(scores_pca)))
[output]
CV=3 인 경우의 PCA 변환된 개별 Fold세트별 정확도: [0.793 0.7958 0.8026]
PCA 변환 데이터 셋 평균 정확도:0.7971
LDA
- 선형 판별 분석법으로 불리며 PCA와 매우 유사
- LDA는 지도학습의 분류에서 사용하기 쉽도록 개별 클래스를 분별할 수 있는 기준을 최대한 유지하면서 차원을 축소
- PCA = 입력 데이터의 변동성이 가장 큰 축을 찾음
- LDA = 입력 데이터의 결정 값 클래스를 최대한으로 분리할 수 있는 축을 찾음
- LDA는 특정 공간상에서 클래스 분리를 최대화하는 축을 클래스 간 분석
- 클래스 내부 분산의 비율을 최대화하는 방식으로 차원을 축소
즉. 클래스 간 분산이 클수록 좋고, 클래스 내부의 분산이 작을수록 좋음
PCA와 LDA를 구하는 단계의 가장 큰 차이점
공분산 행렬이 아닌 한 클래스 간 분산과 클래스 내부 분산 행렬을 생성, 그리고 이 행렬에 기반해 고유벡터를 구하고 입력 데이터를 투영함
LDA STEP
- 클래스 내부와 클래스 간 분산 행렬을 구함, 그리고 이 두개의 행렬은 입력 데이터의 결정 값 클래스별로 개별 피처의 평균 벡터를 기반으로 구함
- 클래스 내부 분산 행렬을 Sw , 클래스 간 분산 행렬을 SB라고 하면 다음 식으로 두 행렬을 고유벡터로 분해 가능
- 고유값이 가장 큰 순으로 K개(LDA변환 차수만큼) 추출
- 고유값이 가장 큰 순으로 추출된 고유벡터를 이용해 새롭게 입력 데이터를 변환
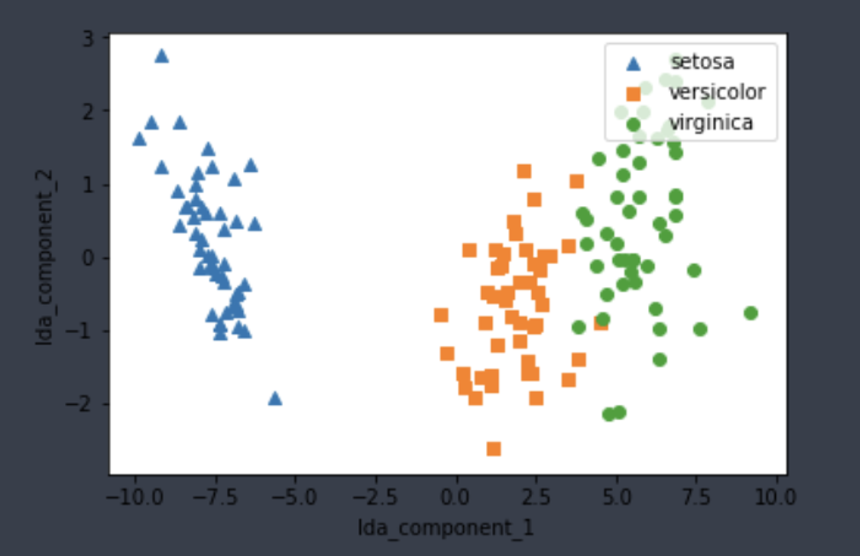
붓꽃 데이터 세트에 LDA 적용하기
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.preprocessing import StandardScaler
from sklearn.datasets import load_iris
iris = load_iris()
iris_scaled = StandardScaler().fit_transform(iris.data)
lda = LinearDiscriminantAnalysis(n_components=2)
# fit()호출 시 target값 입력
lda.fit(iris_scaled, iris.target)
iris_lda = lda.transform(iris_scaled)
print(iris_lda.shape)
[output]
(150, 2)
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
lda_columns=['lda_component_1','lda_component_2']
irisDF_lda = pd.DataFrame(iris_lda,columns=lda_columns)
irisDF_lda['target']=iris.target
#setosa는 세모, versicolor는 네모, virginica는 동그라미로 표현
markers=['^', 's', 'o']
#setosa의 target 값은 0, versicolor는 1, virginica는 2. 각 target 별로 다른 shape으로 scatter plot
for i, marker in enumerate(markers):
x_axis_data = irisDF_lda[irisDF_lda['target']==i]['lda_component_1']
y_axis_data = irisDF_lda[irisDF_lda['target']==i]['lda_component_2']
plt.scatter(x_axis_data, y_axis_data, marker=marker,label=iris.target_names[i])
plt.legend(loc='upper right')
plt.xlabel('lda_component_1')
plt.ylabel('lda_component_2')
plt.show()
SVD
- PCA와 유사한 행렬 분해 기법을 이용
- 차이점
- PCA = 정방행렬만을 고유벡터로 분해 가능
- SVD =정방행렬 뿐 아니라 행과 열의 크기가 다른 행렬에도 적용할 수 있음
- SVD는 특이값 분해로도 불림
- U, V에 속한 벡터는 특이벡터(모든 특이 벡터는 서로 직교하는 성질 존재)
- Σ는 대각 행렬 (행렬의 대각에 위치한 값만 0이 아니고 나머지 위치의 값은 모두 0)
- Σ이 위치한 0이 아닌 값이 바로 행렬 A의 특이값임
- 데이터 세트가 스케일링으로 데이터 중심이 동일해지면 사이킷런의 SVD와 PCA는 동일한 변환 수행
- PCA는 SVD 알고리즘으로 구현됨
- PCA는 밀집 행렬에 대한 변환만 가능, SVD는 희소 행렬에 대한 변환 가능
- SVD는 PCA와 유사하게 컴퓨터 비전 영역에서 이미지 압축을 통한 패턴 인식과 신호 처리 분야에 사용
- 또한 텍스트의 토픽 모델링 기법인 LSA의 기반 알고리즘이기도 함
SVD 실습
[input]
# numpy의 svd 모듈 import
import numpy as np
from numpy.linalg import svd
# 4X4 Random 행렬 a 생성
np.random.seed(121)
a = np.random.randn(4,4)
print(np.round(a, 3))
[output]
[[-0.212 -0.285 -0.574 -0.44 ]
[-0.33 1.184 1.615 0.367]
[-0.014 0.63 1.71 -1.327]
[ 0.402 -0.191 1.404 -1.969]]
[input]
#SVD 핼렬 분해
U, Sigma, Vt = svd(a)
print(U.shape, Sigma.shape, Vt.shape)
print('U matrix:\\n',np.round(U, 3))
print('Sigma Value:\\n',np.round(Sigma, 3))
print('V transpose matrix:\\n',np.round(Vt, 3))
[output]
(4, 4) (4,) (4, 4)
U matrix:
[[-0.079 -0.318 0.867 0.376]
[ 0.383 0.787 0.12 0.469]
[ 0.656 0.022 0.357 -0.664]
[ 0.645 -0.529 -0.328 0.444]]
Sigma Value:
[3.423 2.023 0.463 0.079]
V transpose matrix:
[[ 0.041 0.224 0.786 -0.574]
[-0.2 0.562 0.37 0.712]
[-0.778 0.395 -0.333 -0.357]
[-0.593 -0.692 0.366 0.189]]
[input]
#분해된 행렬들을 이용하여 다시 원행렬로 원복
# Sima를 다시 0 을 포함한 대칭행렬로 변환
Sigma_mat = np.diag(Sigma)
a_ = np.dot(np.dot(U, Sigma_mat), Vt)
print(np.round(a_, 3))
[output]
[[-0.212 -0.285 -0.574 -0.44 ]
[-0.33 1.184 1.615 0.367]
[-0.014 0.63 1.71 -1.327]
[ 0.402 -0.191 1.404 -1.969]]
[input]
#데이터 의존도가 높은 원본 데이터 행렬 생성
a[2] = a[0] + a[1]
a[3] = a[0]
print(np.round(a,3))
[output]
[[-0.212 -0.285 -0.574 -0.44 ]
[-0.33 1.184 1.615 0.367]
[-0.542 0.899 1.041 -0.073]
[-0.212 -0.285 -0.574 -0.44 ]]
[input]
# 다시 SVD를 수행하여 Sigma 값 확인
U, Sigma, Vt = svd(a)
print(U.shape, Sigma.shape, Vt.shape)
print('Sigma Value:\\n',np.round(Sigma,3))
[output]
(4, 4) (4,) (4, 4)
Sigma Value:
[2.663 0.807 0. 0. ]
[input]
# U 행렬의 경우는 Sigma와 내적을 수행하므로 Sigma의 앞 2행에 대응되는 앞 2열만 추출
U_ = U[:, :2]
Sigma_ = np.diag(Sigma[:2])
# V 전치 행렬의 경우는 앞 2행만 추출
Vt_ = Vt[:2]
print(U_.shape, Sigma_.shape, Vt_.shape)
# U, Sigma, Vt의 내적을 수행하며, 다시 원본 행렬 복원
a_ = np.dot(np.dot(U_,Sigma_), Vt_)
print(np.round(a_, 3))
[output]
(4, 2) (2, 2) (2, 4)
[[-0.212 -0.285 -0.574 -0.44 ]
[-0.33 1.184 1.615 0.367]
[-0.542 0.899 1.041 -0.073]
[-0.212 -0.285 -0.574 -0.44 ]]
[input]
#Truncated SVD 를 이용한 행렬 분해
import numpy as np
from scipy.sparse.linalg import svds
from scipy.linalg import svd
# 원본 행렬을 출력하고, SVD를 적용할 경우 U, Sigma, Vt 의 차원 확인
np.random.seed(121)
matrix = np.random.random((6, 6))
print('원본 행렬:\\n',matrix)
U, Sigma, Vt = svd(matrix, full_matrices=False)
print('\\n분해 행렬 차원:',U.shape, Sigma.shape, Vt.shape)
print('\\nSigma값 행렬:', Sigma)
# Truncated SVD로 Sigma 행렬의 특이값을 4개로 하여 Truncated SVD 수행.
num_components = 5
U_tr, Sigma_tr, Vt_tr = svds(matrix, k=num_components)
print('\\nTruncated SVD 분해 행렬 차원:',U_tr.shape, Sigma_tr.shape, Vt_tr.shape)
print('\\nTruncated SVD Sigma값 행렬:', Sigma_tr)
matrix_tr = np.dot(np.dot(U_tr,np.diag(Sigma_tr)), Vt_tr) # output of TruncatedSVD
print('\\nTruncated SVD로 분해 후 복원 행렬:\\n', matrix_tr)
[output]
원본 행렬:
[[0.11133083 0.21076757 0.23296249 0.15194456 0.83017814 0.40791941]
[0.5557906 0.74552394 0.24849976 0.9686594 0.95268418 0.48984885]
[0.01829731 0.85760612 0.40493829 0.62247394 0.29537149 0.92958852]
[0.4056155 0.56730065 0.24575605 0.22573721 0.03827786 0.58098021]
[0.82925331 0.77326256 0.94693849 0.73632338 0.67328275 0.74517176]
[0.51161442 0.46920965 0.6439515 0.82081228 0.14548493 0.01806415]]
분해 행렬 차원: (6, 6) (6,) (6, 6)
Sigma값 행렬: [3.2535007 0.88116505 0.83865238 0.55463089 0.35834824 0.0349925 ]
Truncated SVD 분해 행렬 차원: (6, 5) (5,) (5, 6)
Truncated SVD Sigma값 행렬: [0.35834824 0.55463089 0.83865238 0.88116505 3.2535007 ]
Truncated SVD로 분해 후 복원 행렬:
[[0.11368271 0.19721195 0.23106956 0.15961551 0.82758207 0.41695496]
[0.55500167 0.75007112 0.24913473 0.96608621 0.95355502 0.48681791]
[0.01789183 0.85994318 0.40526464 0.62115143 0.29581906 0.92803075]
[0.40782587 0.55456069 0.24397702 0.23294659 0.035838 0.58947208]
[0.82711496 0.78558742 0.94865955 0.7293489 0.67564311 0.73695659]
[0.5136488 0.45748403 0.64231412 0.82744766 0.14323933 0.0258799 ]]
# 사이킷런 TruncatedSVD 클래스를 이용한 변환
from sklearn.decomposition import TruncatedSVD, PCA
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
%matplotlib inline
iris = load_iris()
iris_ftrs = iris.data
# 2개의 주요 component로 TruncatedSVD 변환
tsvd = TruncatedSVD(n_components=2)
tsvd.fit(iris_ftrs)
iris_tsvd = tsvd.transform(iris_ftrs)
# Scatter plot 2차원으로 TruncatedSVD 변환 된 데이터 표현. 품종은 색깔로 구분
plt.scatter(x=iris_tsvd[:,0], y= iris_tsvd[:,1], c= iris.target)
plt.xlabel('TruncatedSVD Component 1')
plt.ylabel('TruncatedSVD Component 2')

from sklearn.preprocessing import StandardScaler
# iris 데이터를 StandardScaler로 변환
scaler = StandardScaler()
iris_scaled = scaler.fit_transform(iris_ftrs)
# 스케일링된 데이터를 기반으로 TruncatedSVD 변환 수행
tsvd = TruncatedSVD(n_components=2)
tsvd.fit(iris_scaled)
iris_tsvd = tsvd.transform(iris_scaled)
# 스케일링된 데이터를 기반으로 PCA 변환 수행
pca = PCA(n_components=2)
pca.fit(iris_scaled)
iris_pca = pca.transform(iris_scaled)
# TruncatedSVD 변환 데이터를 왼쪽에, PCA변환 데이터를 오른쪽에 표현
fig, (ax1, ax2) = plt.subplots(figsize=(9,4), ncols=2)
ax1.scatter(x=iris_tsvd[:,0], y= iris_tsvd[:,1], c= iris.target)
ax2.scatter(x=iris_pca[:,0], y= iris_pca[:,1], c= iris.target)
ax1.set_title('Truncated SVD Transformed')
ax2.set_title('PCA Transformed')

NMF
- Truncated SVD와 같이 낮은 랭크를 통한 행렬 근사 방식의 변형
- 원본 행렬 내의 모든 원소 값이 모두 양수라는 게 보장되면 더 간단하게 두개의 기반 양수 행렬로 분해될 수 있는 기법
- 행렬 분해는 일반적으로 SVD와 같은 행렬 분해 기법을 통칭하는 것
- 행렬 분해 시작고 넓은 행렬 H로 분해
- 분해 행렬 W는 원본 행에 대해서 이 잠재 요소 값이 얼마나 되는지에 대응
- 분해 행렬 H는 이 잠재 요소가 원본 열로 어떻게 구성 됐는지 나타내는 행렬
- 일반적으로 길고 가는 행렬 w
- SVD와 유사하게 차원 축소를 통한 잠재 요소 도출 (이미지 변환 및 압축, 텍스트의 토픽 도출 영역에서 사용….→이미지 압축을 통한 패턴 인식, 텍스트의 토픽 모델링 기법, 문서 유사도 및 클러스터링
- 또한 영화 추천과 같은 추천 영역에 활발하게 적용
from sklearn.decomposition import NMF
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
%matplotlib inline
iris = load_iris()
iris_ftrs = iris.data
nmf = NMF(n_components=2)
nmf.fit(iris_ftrs)
iris_nmf = nmf.transform(iris_ftrs)
plt.scatter(x=iris_nmf[:,0], y= iris_nmf[:,1], c= iris.target)
plt.xlabel('NMF Component 1')
plt.ylabel('NMF Component 2')

차원 축소 정리
- 차원 축소는 단순히 피처의 개수를 줄이는 개념보다는 데이터를 잘 설명할 수 있는 잠재적인 요소를 추출
- PCA
- 입력 데이터의 변동성이 가장 큰 축을 구함
- 다시 이축에 직각인 축을 반복적으로 축소하려는 차원 개수만큼 구함
- 입력 데이터를 이 축들에 투영해 차원을 축소
이를 위해 입력 데이터의 공분산 행렬을 기반으로 고유 벡터를 생성 후 이렇게 구한 고유 벡터에 입력 데이터를 선형 변환하는 방식
- LDA
- PCA와 매우 유사한 방식이며, PCA가 입력 데이터 변동성의 가장 큰 축을 찾는데 반해 LDA는 입력 데이터의 결정 값 클래스를 최대한으로 분리할 수 있는 축을 찾는 방식
- SVD, NMF
- 매우 많은 피처 데이터를 가진 고차원 행렬을 두 개의 저차원 행렬로 분리하는 행렬 분해 기법
- 특히 이러한 행렬 분해를 수행하면서 원본 행렬에서 잠재된 요소를 추출하기 때문에 토픽 모델링이나 추천 시스템에사 활발히 사용됨